Post 43: Leyes de escala y modelos de lenguaje de proteinas 💸
Published:
Hoy martes, martes de solo China podría haber logrado 😬
Los modelos de lenguaje son los algoritmos que hacen que IAs como ChatGPT funcionen. Estos algoritmos aprenden múltiples temas a la vez (medicina, arquitectura, etc.). Para que esto sea posible es necesario considerar dos aspectos: la cantidad de datos usados en el entrenamiento y el número de conexiones neuronales (AKA el tamaño del modelo).
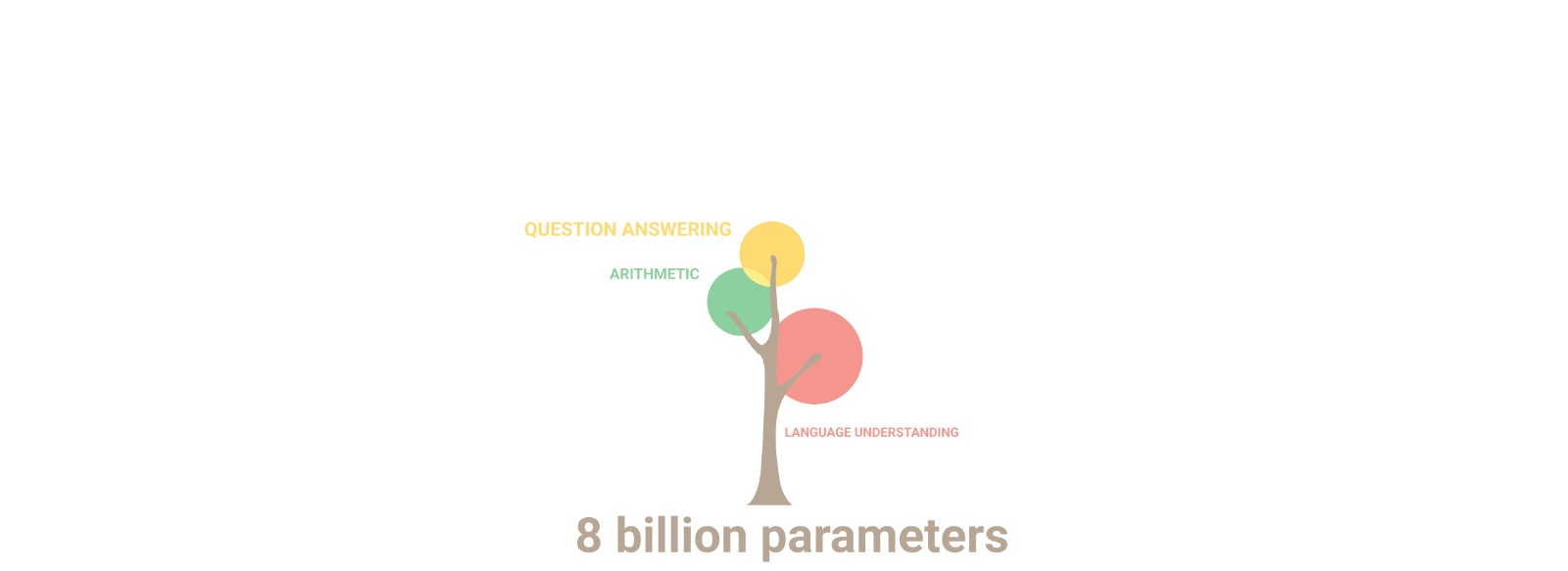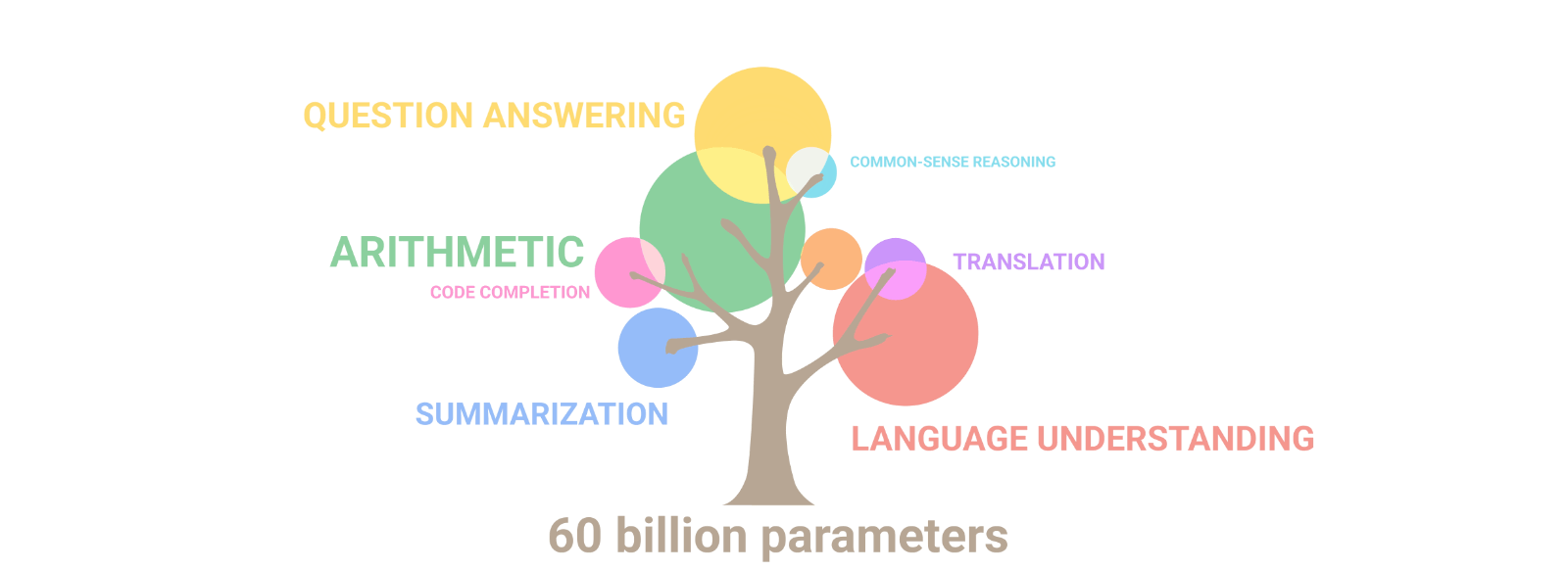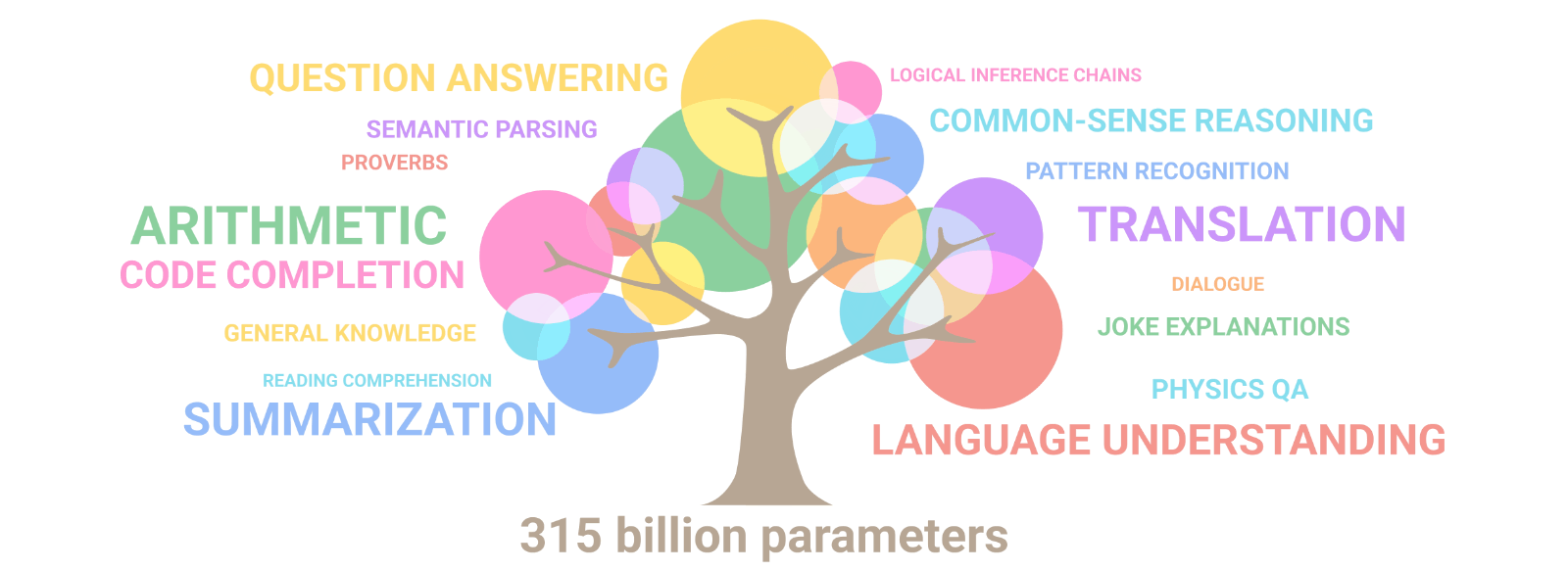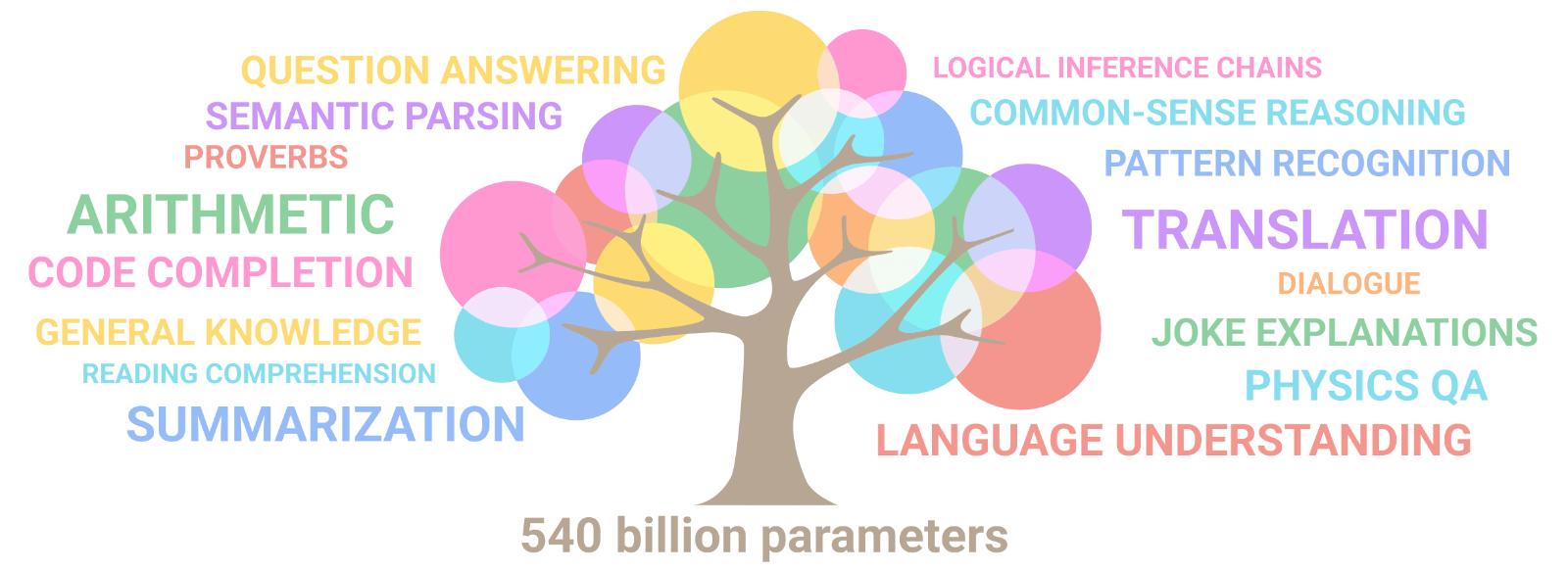
Las capacidades de estos modelos siguen unas “leyes de escala” que dicen que: a medida que aumenta el tamaño del modelo, la IA aprende mejor los temas y, además, aparecen otros nuevos. Como lo ejemplifican las ramas del árbol y su tamaño. Por ejemplo: al duplicar el tamaño del modelo, la IA pasa de saber biología básica a saber genética. Por esta razón, es que hay IAs cada vez más grandes. Pero ¿Qué pasaría si en vez de texto se usaran proteínas?
En 2019 aparecieron los primeros modelos de lenguaje de proteínas (más detalles en las refs 1 y 2). Estos modelos aprenden aspectos de las proteínas como su ubicación subcelular, tipo de plegamiento, función, etc. “ESM-2”, entrenada por FaceBook, era la IA de proteínas más grande hasta ahora y con ella predijeron la estructura de 600 millones de proteínas (ref 3). Pero eso cambio hace apenas un par de semanas.
La empresa china BioMap ha presentado su IA llamada “xTrimoPGLM”, la cual es ~7 veces más grande que la IA de FaceBook. Además, xTrimoPGLM es la primera IA en su tipo en el mundo de las proteínas, pues es tan grande como otras IAs de texto famosas como lo es GPT-3. Al compararla con otras IA, xTrimoPGLM ha conseguido un mejor desempeño en múltiples tareas predictivas relacionadas a las propiedades de las proteínas y también es capaz de generar secuencias. Es de esperar que nuevas estrategias deriven de esta IA en los siguientes años, sin embargo, aún no es seguro que BioMap haga “open source” este modelo y tenderemos que esperar un poco más.
Dato curioso: el entrenamiento de xTrimoPGLM empezó el 18 de Enero y “terminó” el 30 de Junio. El preprint se publicó el 14 de Julio. Lo cual significa que el equipo chino tardó 14 días en hacer todos los análisis presentados ggg
Refs:
