Post 6: Modelos de lenguaje de proteínas 💻
Published:
Previamente escribí cómo pensar a las proteínas como lenguajes. Aquí, más detalles de uno de estos lenguajes de proteínas (ESM-1b).
Modelar la biología es difícil, pues no suele ser aditiva, si no que tiende a ser sinérgica, emergente y contexto dependiente. Dado que los sistemas biológicos son más que la suma de sus partes, hacer predicciones útiles solo a partir de ellas es extremamente complicado. Y empeora si consideras que evolucionan en el tiempo y que lo que vemos hoy, es solo un fragmento diminuto de la biodiversidad que ha existido en la Tierra (la propiedad histórica de la biología). Por lo que un modelo que logre capturar tanta información parecía imposible de conseguir hasta hace poco.
Dentro del área de Inteligencia artificial, el procesamiento de lenguaje natural es una estrategia que ha permitido “semantizar” toneladas de información. La forma más fácil de entender estos modelos (por ejemplo, GPT-J), es mediante la generación de texto coherente dado una frase como base. En biología, esto se refleja de muchas maneras, por ejemplo, aprendiendo a agrupar proteínas de acuerdo con su estructura secundaria (F1).
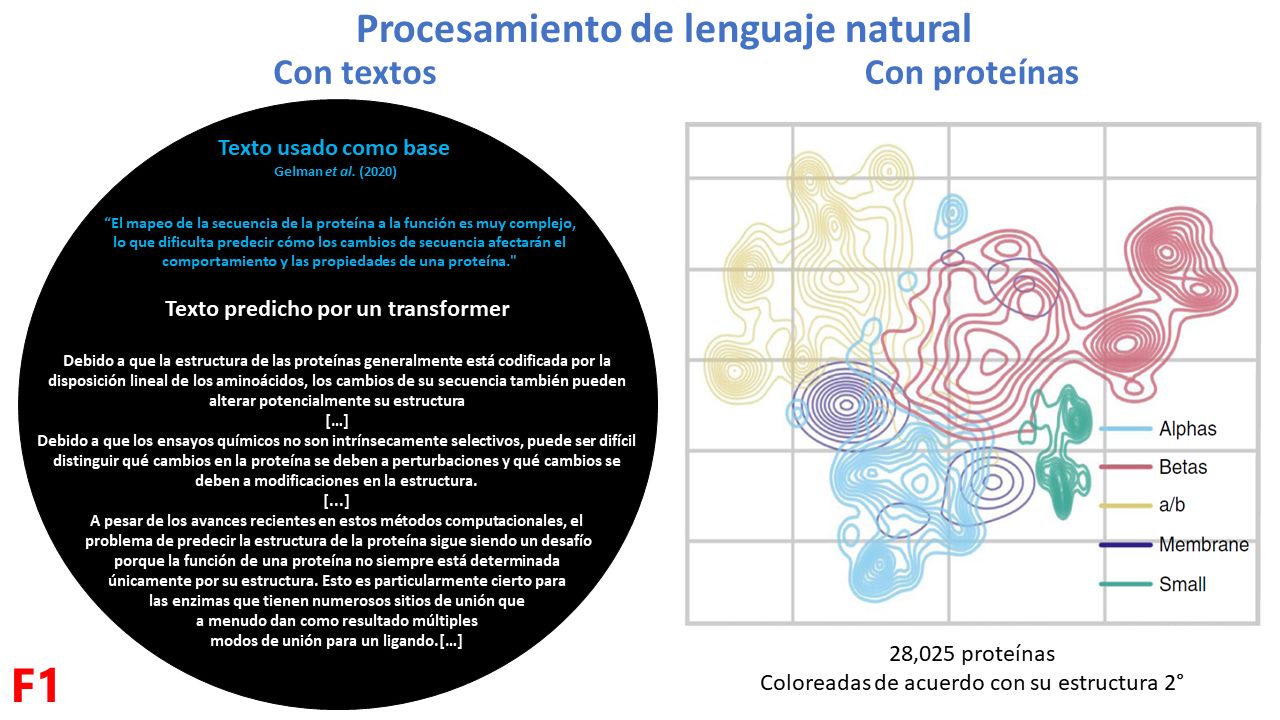
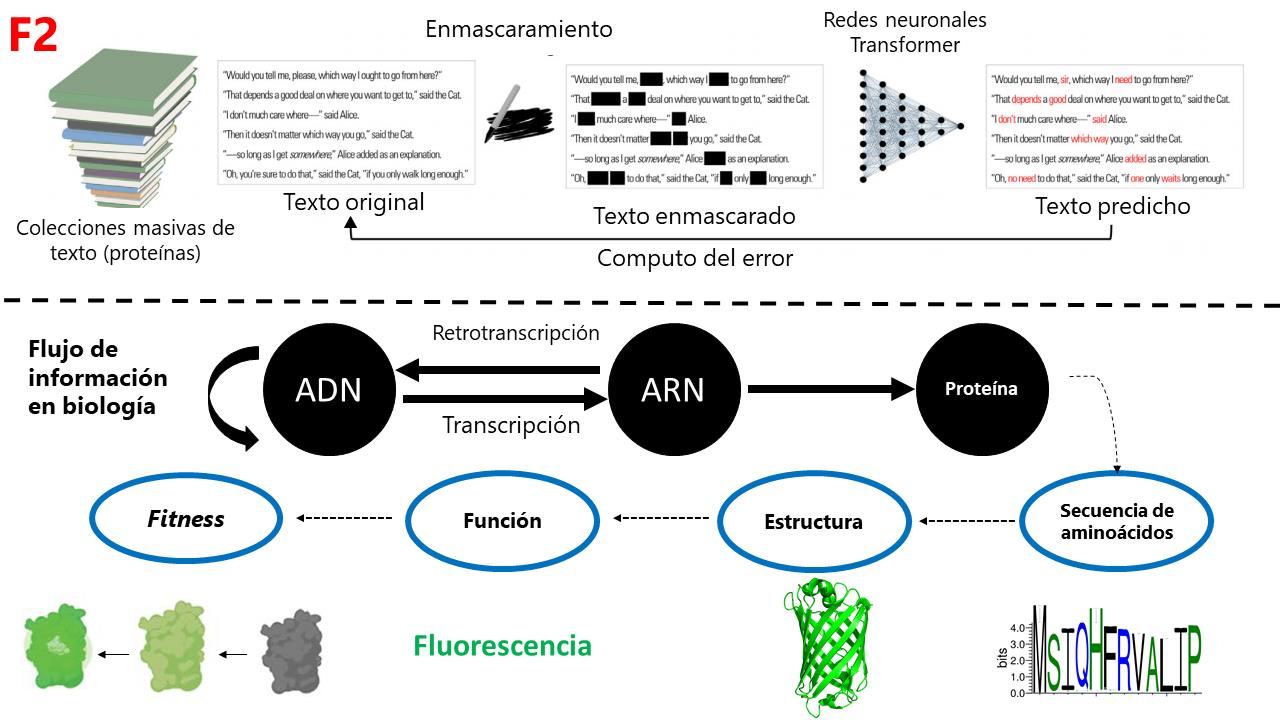
ESM-1b (Evolutionary Scale Modeling) es un modelo que ha aprendido biología al observar 250 millones de proteínas. Su premisa es que las proteínas que vemos hoy son también “fósiles informacionales” que, al haber sido moldeadas por la evolución, contienen “reglas” de lo que es más biofísicamente probable. ESM-1b aprendió biología al tomar cada una de las proteínas, enmascarar algunos de sus aminoácidos y comparar está proteína enmascarada contra su versión original para computar cuáles son las combinaciones de aminoácidos más probables. Lo cual, le permitió “destilar” propiedades emergentes como la estructura y función de las proteínas, e incluso el grado de funcionalidad (o fitness) de las mismas (F2).
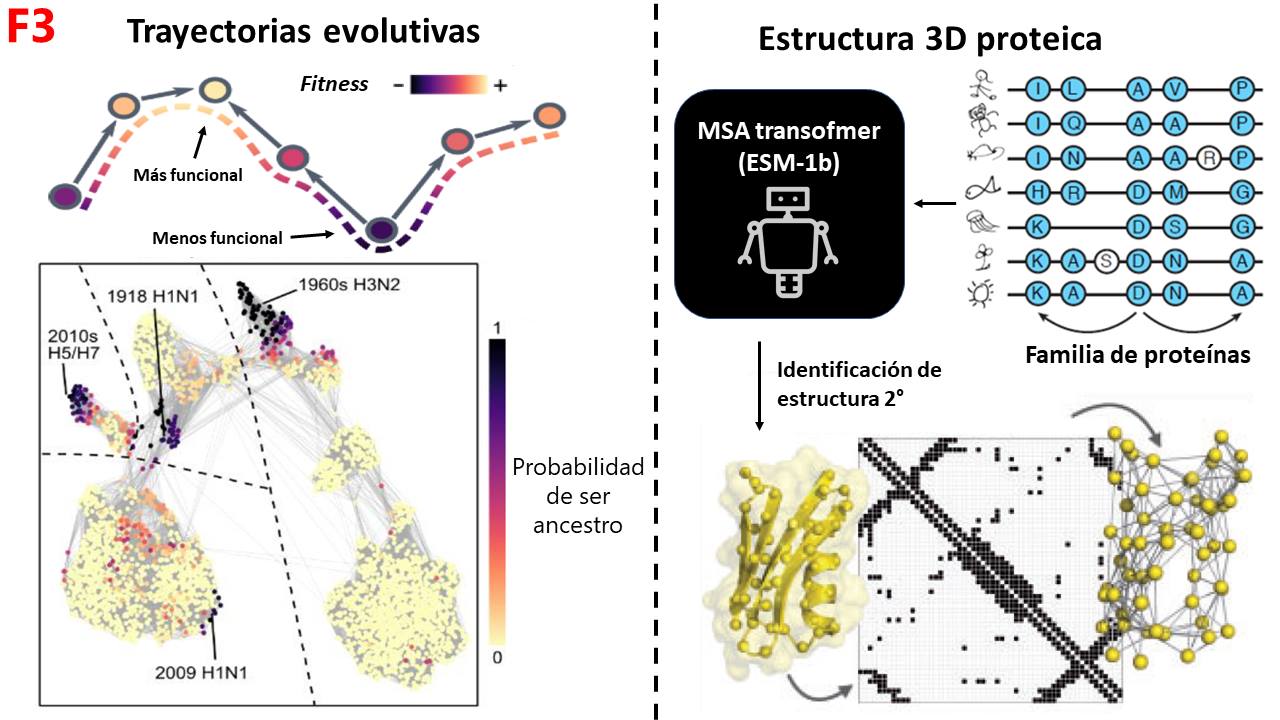
ESM-1b puede usarse para predecir el fitness de las proteínas y trazar trayectorias evolutivas de menos a más funcionales; y luego, asignar cuales proteínas fueron probablemente los ancestros de todo un conjunto. Si esto lo aplicamos al virus de la influenza, podemos ver qué ESM-1b identifica como puntos de origen (ancestros) a proteínas que tienen origen en pandemias, como las del siglo XX o la del 2009. Permitiendo modelar parte de la dinámica de las pandemias. ESM-1b, también puede usarse para identificar la estructura 3D de las proteínas (MSA-Transformer). Para ello se hace un entrenamiento especializado con proteínas de la misma familia para aprender a distinguir que regiones de aminoácidos son más probables de tener cierta estructura secundaria y así identificar el plegamiento 3D completo. De hecho, está misma estrategia es usada por AlphaFold2 con un módulo llamado Evoformer (F3).
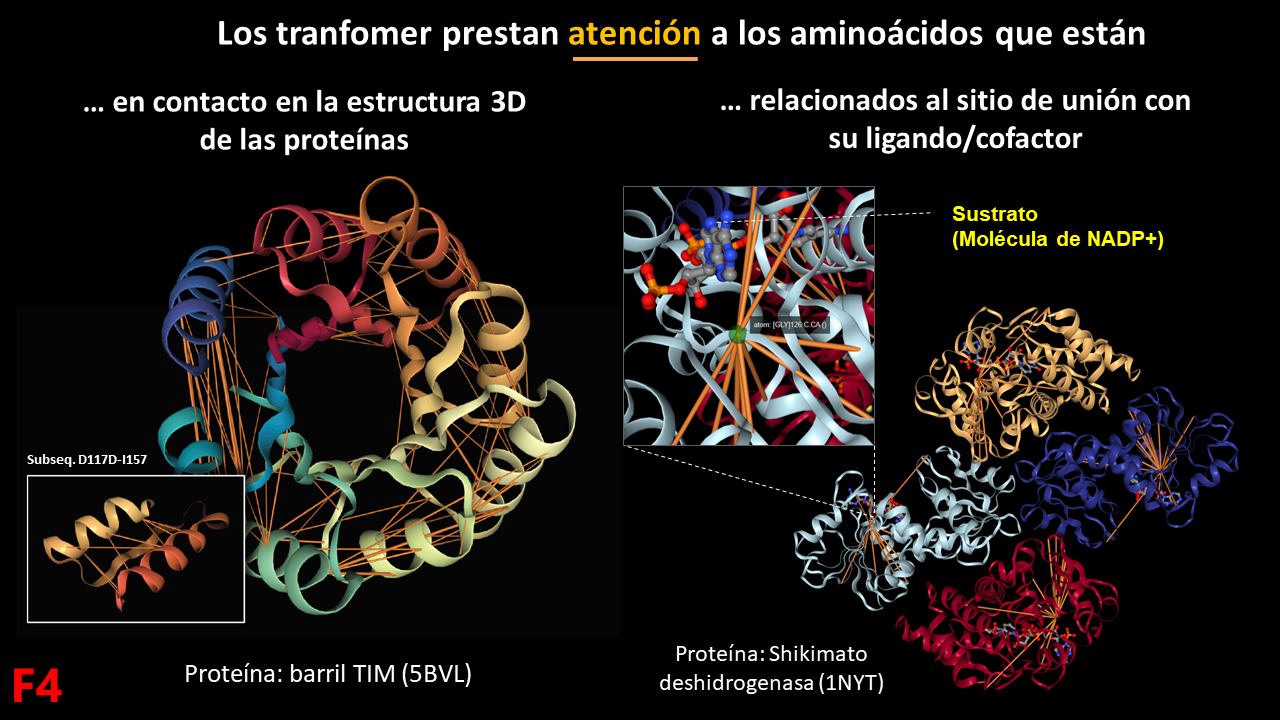
En el corazón de ESM-1b hay una operación matemática conocida como “Atención”, la cual caracteriza a un tipo de redes neuronales conocidas como “Transformers”. La atención, le permite a los transformes identificar las partes más relevantes de una secuencia de información. Si la vemos en las proteínas, los aminoácidos que tienen más atención entre si son aquellos que están en contacto 3D en la proteína, e incluso, hay mucha atención a los aminoácidos que están relacionados al sitio de unión al sustrato en el caso de las enzimas. Lo cual demuestra que durante el entrenamiento de ESM-1b con millones de proteínas, en cada caso, presta atención a los aminoácidos más importantes de la estructura y función.
Las aplicaciones se ESM-1b y demás transformes apenas empiezan. De hecho, existe otro modelo (ProGen) que ha aprendido a sintetizar proteínas solo diciéndole el tipo de proteína que quieres (p. ej. una hidrolasa de Homo sapiens de 222 aminoácidos se longitud). Lo que nos espera en esta década dentro del área del procesamiento del lenguaje natural aplicado en biología es emocionante! Todo lo contrario a lo que nos depara el cambió climático …
No quisiera ir a ningún lado si mi increíble toalla.
Refs:
Articulo base de ESM-1b
Aplicaciones a la estructura 3D de las proteínas
- Transformer protein language models are unsupervised structure learners
- BERTology Meets Biology: Interpreting Attention in Protein Language Models
Aplicaciones a la función de 3D de las proteínas
- Language models enable zero-shot prediction of the effects of mutations on protein function
- Evolutionary velocity with protein language models
- Evotuning protocols for Transformer-based variant effect prediction on multi-domain proteins
Modelo ProGen
