Post 4: ¿Cómo pensar a las proteínas? 🤔
Published:
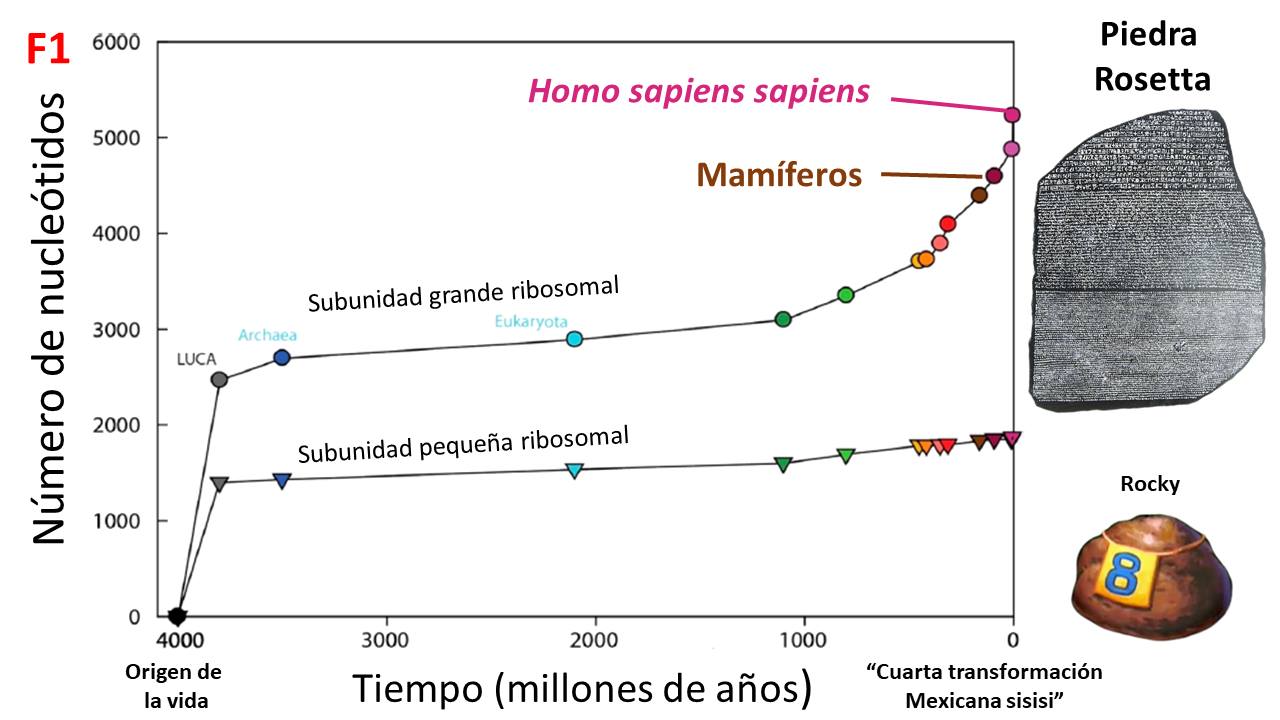
1) Como relojes: algunos darán la hora mientras que otros hasta segundos. Esta carga de información de las proteínas se debe a los patrones en como acumulan mutaciones en sus genes, las cuales se reflejan en aminoácidos sujetos a distintas fuerzas evolutivas. Esta idea nos ha servido para datar el origen de la vida en la Tierra, por ejemplo, usando proteínas ribosomales (F1).
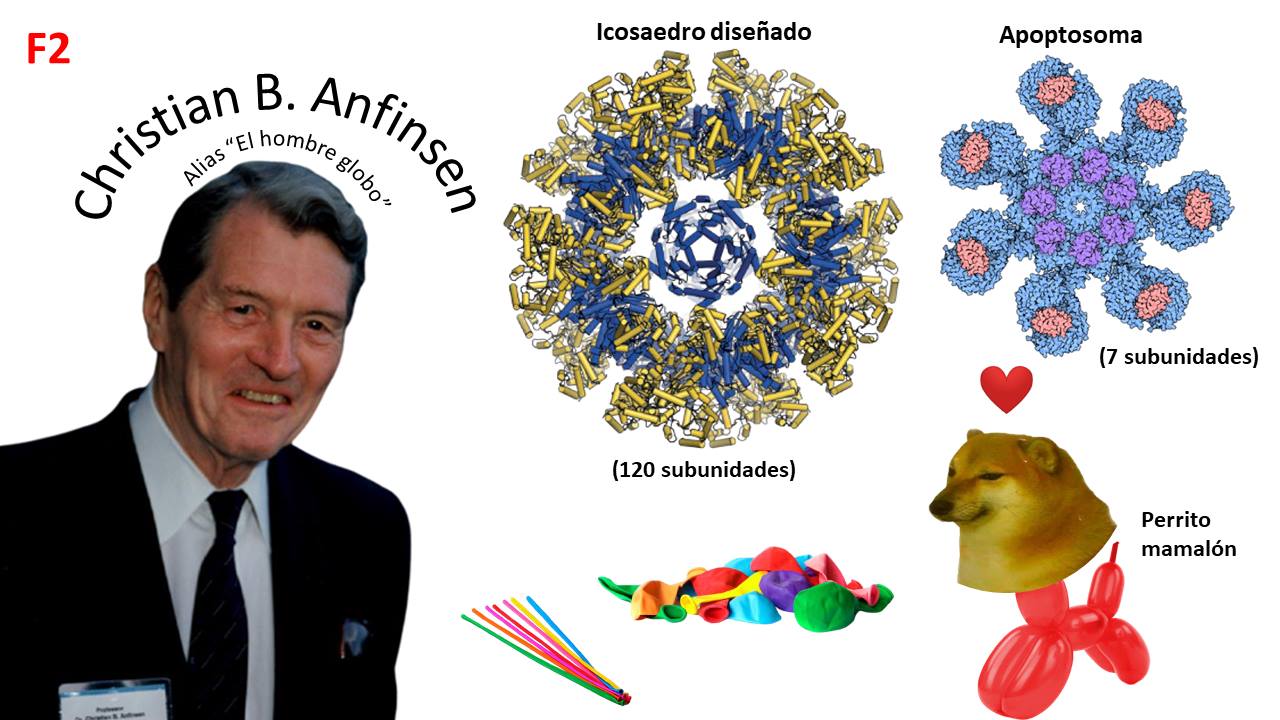
2) Como globos: algunos permiten hacer formas raras como “apoptosomas”, otros es mejor ni tocarlos porque explotan. Así como los globos toman forma a partir de su envase y aire, las proteínas se pliegan a partir de la interacción entre sus aminoácidos y la termodinámica que ocurre en el disolvente (awa). Christian Anfinsen propuso que toda la información para que una proteína se pliegue está contenida en sus aminoácidos. Hoy esta idea se refleja en AlphaFold, un conjunto de redes neuronales que predicen la estructura 3D de una proteína a buena resolución.
3) Como lenguajes: las proteínas no solo tienen información temporal y estructural codificada, también funcional. Sus aminoácidos tienen una semántica. En este sentido, se parecen a la piedra Rosetta, la cual tenía un mensaje encriptado en 3 lenguajes diferentes. Las funciones y el que “tan bien” desempeñan las proteínas dependen tanto de la identidad y posición 3D de los aminoácidos.
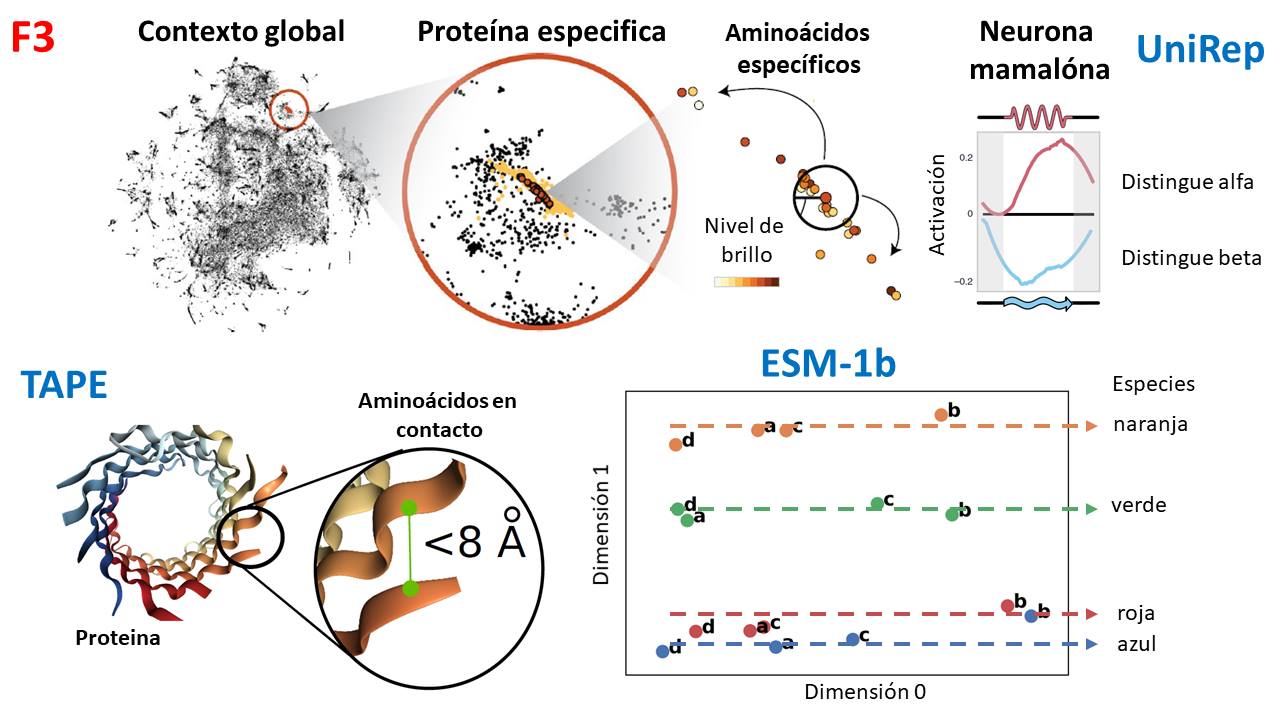
Usando redes neuronales y métodos de procesamiento de lenguaje natural (como BERT), podemos entrenar modelos que aprendan a codificar la semántica de los aminoácidos y después, pedirles a estos modelos que nos den información de la historia evolutiva, estructura 3D y función de las proteínas. Incluso, estos modelos pueden crear proteínas artificiales bajo limites biofísicos que uno les indique. Hay muchos “lenguajes neuronales de proteínas”, pero de ellos me llaman la atención estos (F3):
- “UniRep”: desarrollado en Harvard, se entrenó con 24 millones de proteínas y se encontró una neurona capaz de distinguir entre los tipos de plegamientos de proteínas (alfa y beta). Además de que es posible afinar el modelo para preguntarle que aminoácidos mejorarían un rasgo de una proteína, por ejemplo, el brillo una proteína fluorescente.
- “TAPE”: desarrollado en Google y Berkley, proponen una serie de métricas para evaluar los lenguajes neuronales de proteínas y además, entrenan un tipo de redes neuronales conocidas como “Transformers” con 31 millones de proteínas y se encontró que es capaz de predecir como se pliegan las proteínas para mejorar su estabilidad.
- “ESM-1b:” desarrollado en FaceBook y universidades estadounidenses, es uno de los dos modelos mas grandes hasta ahora que se entreno con 250 millones de proteínas y es capaz de distinguir qué tipo de proteínas (naranja, verde, rojo y azul) corresponde a qué tipo de especies (A, B, C y D).
Lo importante es que estas propiedades se aprendieron SOLO a partir de las secuencias de aminoácidos, ningún otro tipo de información se uso para entrenar. Es decir, las redes aprendieron la semántica y aspectos emergentes de las proteínas, como sus relaciones evolutivas. Lo cual es increíble.
Coman frutas y verduras.
Refs:
- Pensar a las proteínas como relojes (evolución):
- Pensar a las proteínas como globos (biofísica):
- introducción a lenguajes de proteínas:
- Redes neuronales:
