Post 52: La evolución de AlphaFold 🕦
Published:
AlphaFold (AF) es la inteligencia artificial (IA) más famosa en el mundo científico debido a que fue la primera en resolver el reto de predecir la estructura atómica de las proteínas, aunque no de todas las internaciones entre biomoléculas. Sin embargo, las ideas de cada una de sus tres versiones son radicalmente distintas.
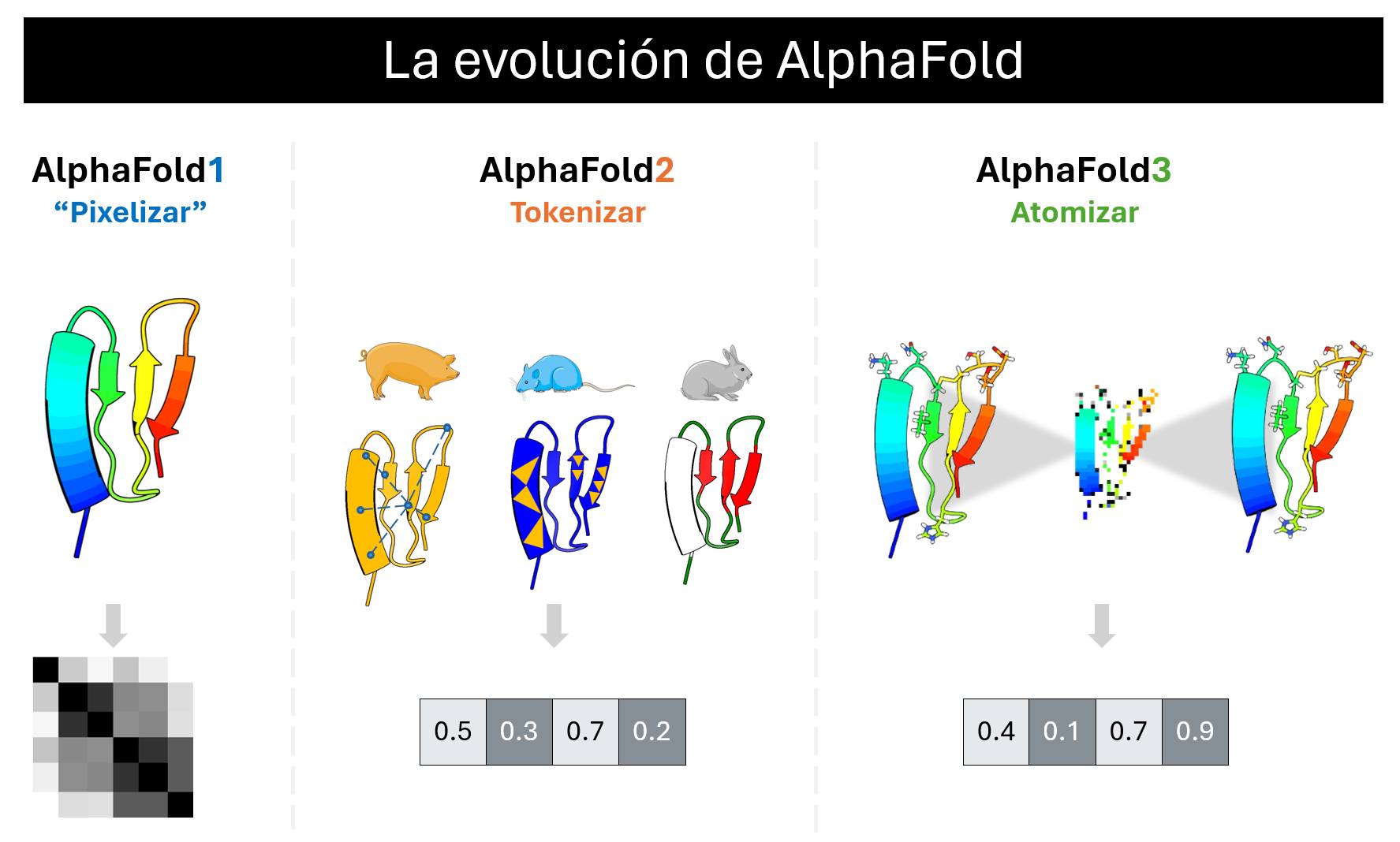
La innovación detrás de AF1 fue “pixelizar” a las proteínas, es decir, representarlas como imágenes que capturan las distancias entre átomos. Esta forma de representar a las proteínas permitió analizarlas con una IA especializada en imágenes, y aunque no lograron superar el reto, si superaron al resto de competidores por mucho.
En AF2 se dieron cuenta que AF1 no sería suficiente, y tocó desechar todo y hacer algo nuevo. La innovación detrás de AF2 fue el “tokenizar” a las proteínas, es decir, partirlas en cachitos para permitir que una IA de forma autónoma aprendiera a representar a las proteínas como vectores numéricos. Esto es muy distinto, pues en AF1 nosotros le indicamos a la IA como representar a las proteínas, pero en AF2 es la misma IA la que aprendió a representarlas de la forma más óptima posible. Para que AF2 hiciera esto, fue necesario escribir algoritmos fuertemente inspirados en la evolución, bioquímica y biofísica de las proteínas, por lo que es un algoritmo muy apreciado por lo biólogos.
Sin embargo, AF2 fue víctima de su propio éxito. Al tener tantas bases bioinspiradas en su algoritmo, no generaliza al inmenso mundo de interacciones entre biomoléculas. Así que tocó desechar todo y empezar de cero, otra vez. La innovación detrás de AF3 fue “atomizar” a las proteínas, es decir, ya no solo partirlas en cachitos, si no representarlas como la mínima unidad posible de las moléculas, el átomo. Al atomizar el algoritmo, fue posible descartar prácticamente todas las bases biológicas que se habían escrito para AF2, permitiendo que la IA fuera la que aprendiera dichas bases de forma autónoma. Aqui nuevamente, la proteínas se representan como vectores, pero ahora mediante un proceso de prueba y error donde Ia IA reconstruye los átomos de las proteínas.
En realidad, ninguna de las innovaciones de AF son realmente originales. Todas fueron ideas previamente exploradas, pero los científicos de Google lograron integrarlas de una forma muy bien pensada tal que, junto con su gran capacidad de cómputo, pudieran entrenar a cada una de las versiones de AF. Además, es bastante sorprendente ver como se desecharon la mayoría de las bases biológicas para favorecer cada vez más la autonomía de la IA; lo cual pese a servir bastante bien, parece ir en contra a lo que usualmente hacemos en la ciencia: comprender algo para luego pasar a sus aplicaciones.
Así que nuevas formas de analizar y/o abordar las cosas abren nuevos caminos … a veces bastante interesantes.
Referencias:
- (sobre el cambio de paradigma de AF2 a AF3) The principle of uncertainty in biology: Will machine learning/artificial intelligence lead to the end of mechanistic studies?
- (Sobre el paso de AF1 a AF2) Starting at Go: Protein structure prediction succumbs to machine learning
- (sobre las bases biológicas de AF2) Protein structure prediction by AlphaFold2: are attention and symmetries all you need?
Y muchos otros post previos sobre AF:
- Un plegamiento muy extraño
- 200 Millones de estructruas nuevas!
- Un año después de AlphaFold2
- ¿Cuánto costó entrenar AlphaFold2?
- AlphaFold2, articulo científico más famoso
- Como aprendió AlphaFold2 a modelar las proteínas?
Addendum:
Quieren saber que existió antes de AlphaFold? Aqui una muy completa historia al respecto por parte de quantamagazine. Interesantes son las citas de varios de los personajes implicados, como Janet Thornton:
- Other AlphaFold competitors, including Meta, crafted their own algorithms […]. However […], no one has been able to match AlphaFold’s accuracy so far, Thornton said “I’m sure they will, but I think getting another … AlphaFold moment like that will be very difficult.”
- How AI Revolutionized Protein Science
- Quien es Janet Thornton?
AF2 es el momento “AlexNet” de la intersección entre la bio y la IA. Es decir, aquel momento que convenció a la gente a voltear a ver/usar estas herramientas. Y tan cierto es que basta con ver las estadisticas del articulo. Ademas ahora hay un boom de startups de bio-ia-generativa (generatebiomedicines, profluent, DynoTx, Nabla Bio, etc) asi como una considerable cantidad de premios relacionados a DeepMind o al laboratorio de Baker
En lo particular, me desagrada bastante su naturaleza generativa de AF3 porque me da desconfianza respecto a AF2. Además, no hay que confundir el reto de la CASP (i.e. predecir la estructura) con el comportamiento dinámico de las proteinas y su “folding” como para explicar su estructura-funcion.
