Post 46: Una IA que crea proteínas de la nada 🎩🪄
Published:
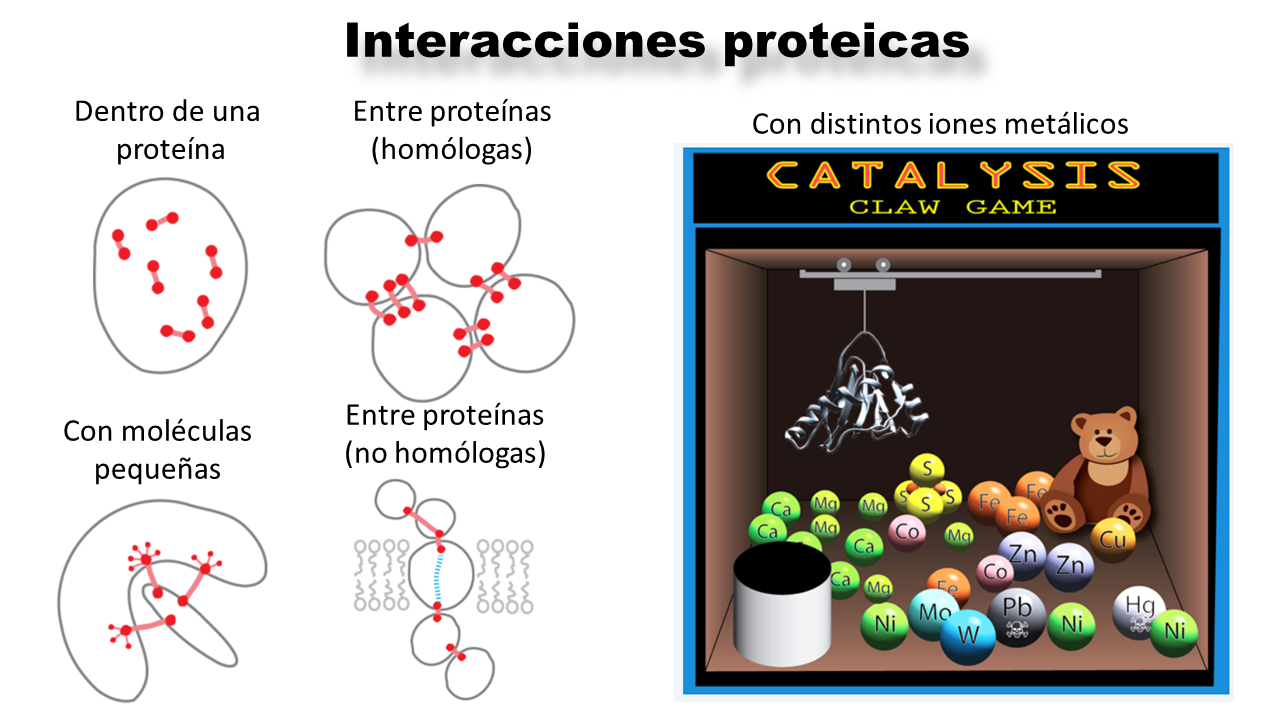
Para que una proteína funcione primero debe plegarse. Para que eso ocurra, determinados aminoácidos deben entrar en contacto y así favorecer las condiciones energéticas y geométricas necesarias. Sin embargo, las proteínas raramente actúan solas; usualmente interactúan entre ellas, lo hacen también con el ADN/ARN, con otras moléculas pequeñas (como los antibióticos) e incluso con iones metálicos (como en la fotosíntesis). Cuando una proteína interactúa de estas formas, le llamamos complejo proteico.
AlphaFold2 ha revolucionado la ciencia de proteínas, pero su gran debilidad es que no puede predecir los enlaces químicos que se dan en un complejo proteico (de hecho, tampoco sabe modelar interacciones como los puentes de disulfuro). Para intentar resolver esto, en el laboratorio de David Baker han entrenado RoseTTAFold All-Atom (RFAA), una nueva IA capaz de crear proteínas desde cero.
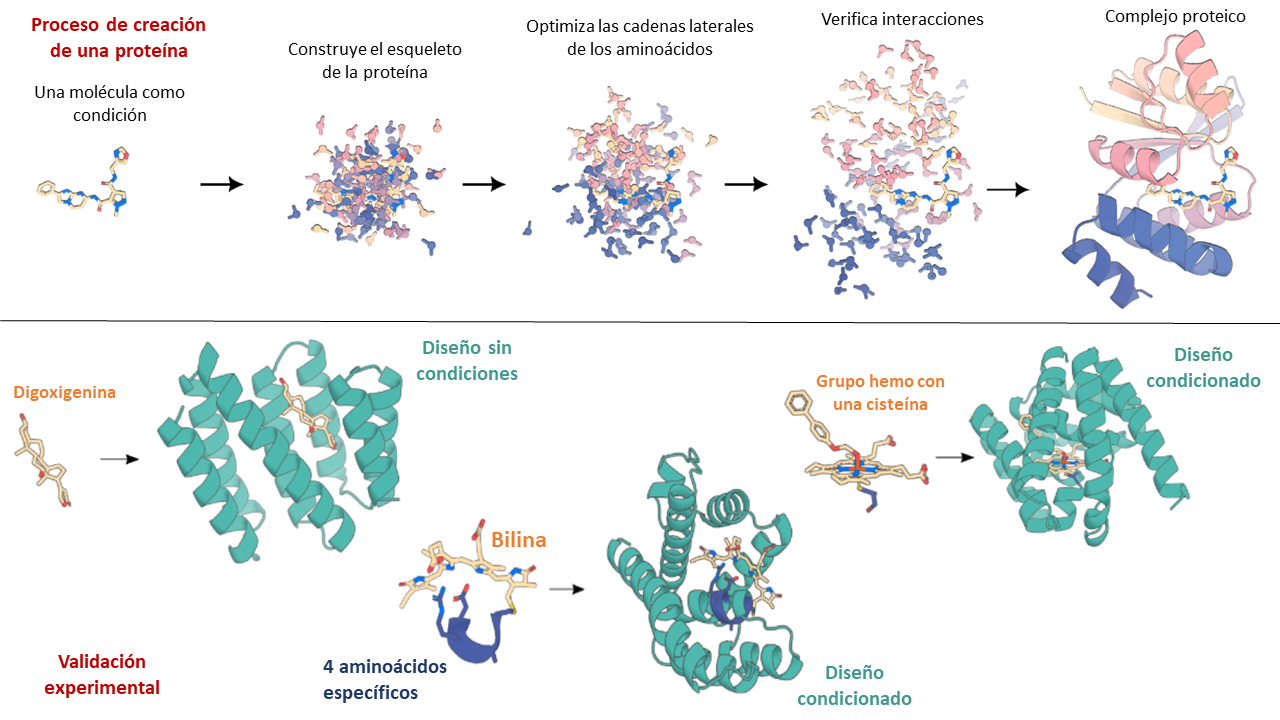
RFAA fue entrenada prestando atención a los enlaces químicos que se establecen cuando las proteínas interactúan. Para ello, usaron muchos complejos proteicos, y además, representaron átomo por átomo a las regiones que están interactuando (y no aminoácido por aminoácido, como normalmente se hace). Con esto logran hacer que dado una molécula o interacción especifica, la IA construya una proteína que satisfaga dicha interacción, como lo podría ser la de una enzima y un antibiótico. Es decir, es una IA que construye sobre un molde (interacción) especifico.
Las capacidades esta IA para crear nuevas proteínas han sido demostradas al comprobar experimentalmente tres casos donde las proteínas logran plegarse y cumplir su función. En el primer caso dejaron que la IA creara una proteína desde cero que fuera capaz de unirse a un esteroide llamado digoxigenina (usado para enfermedades cardiacas). En el segundo caso le pidieron crear una proteína que se uniera a un grupo hemo (como el de la hemoglobina) alrededor de una cisteína. Y en el tercer caso le pidieron una proteína que se uniera mediante cuatro aminoácidos específicos a unos pigmentos llamados bilinas que están involucrados en la fotosíntesis.
Lo más interesante es que las proteínas creadas por IA no se parecen a ninguna proteína usada en el entrenamiento, lo cual sugiere que la IA aprendió nociones de la física y geometría de las interacciones proteicas. Sin embargo, los autores reconocen que es necesario mejorar su desempeño usando más datos de entrenamiento. Y por el momento, no han liberado su código, aunque podemos esperarlo dentro de unos 6/8 meses cuando se publique el articulo oficialmente.
Referencias: