Post 3: Ingeniería de proteínas basada en inteligencia artificial 🤖
Published:
Me gustaría decir que la biología ha pasado por 3 grandes transformaciones analíticas:
- una “molécularizacion” con la caracterización de la estructura del ADN.
- una digitalización con a las tecnologías de secuenciación masiva.
- una “semántizacion” con el computo masivo de datos.
Para dar contexto a los datos se usan técnicas de aprendizaje automático, en donde las redes neuronales han sido las más famosas (F1). Dentro de la gran diversidad de datos biológicos que se pueden analizar, las estructuras 3D de proteínas son una fuente rica de información que puede ser aprovechada para entender su función y mejorar sus capacidades. 
Para ello, el laboratorio de Russ tomó las estructuras proteicas y las partió en pequeños cachitos que representan los microambientes químicos de los átomos de C, O, N y S con el fin de predecir regiones dentro de la proteína que son propensas a cambiar. Unos años después, el equipo de Ross retomaría la red neuronal de Russ y la mejoraron al añadir informacion mas detallada, por ejemplo, considerando todos los H.
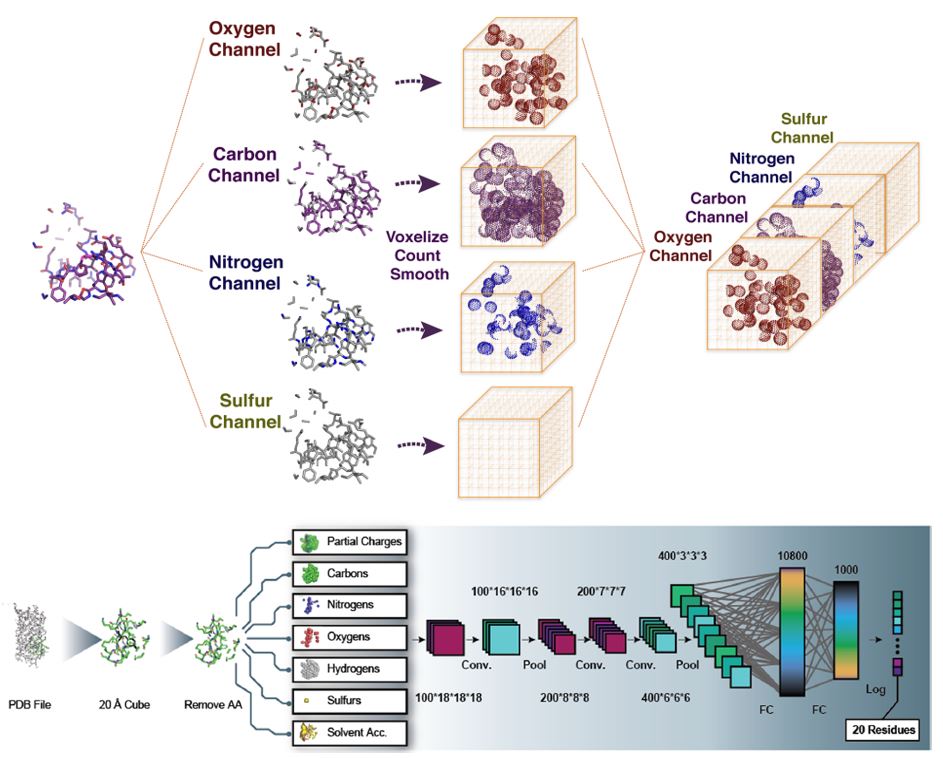
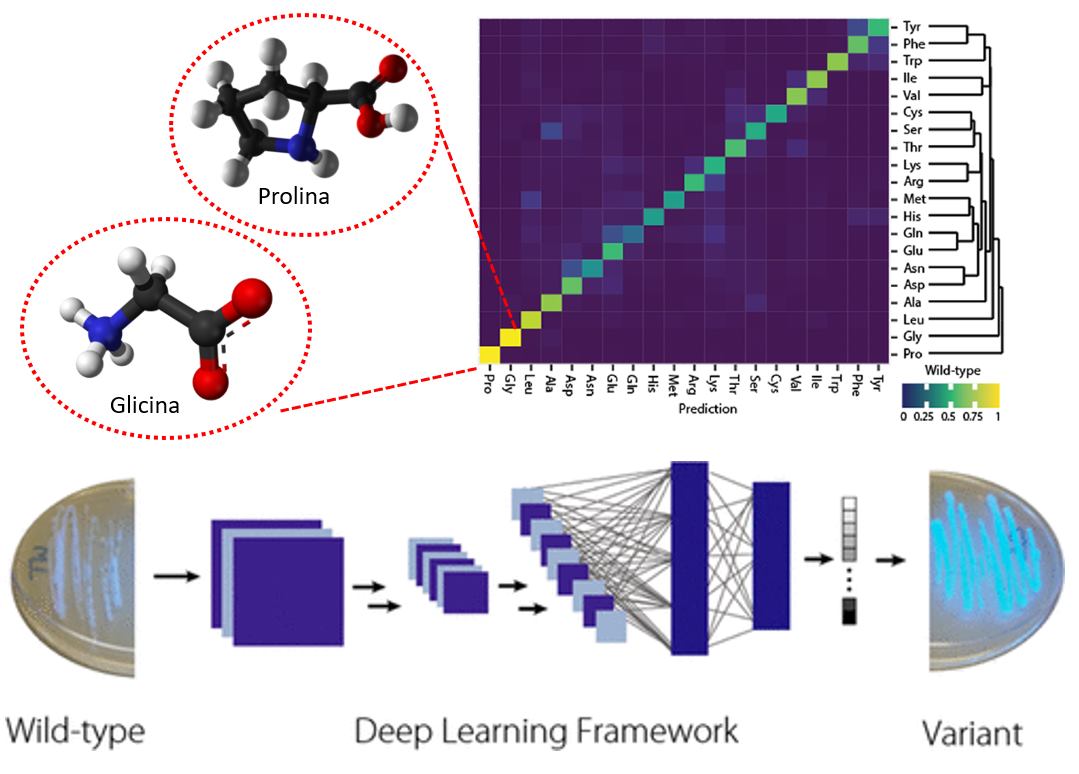
Con esto, la red aprendió a modelar los microambientes químicos dentro de las proteínas y recapitula ciertas propiedades ya conocidas de los aminoácidos, como que la Prolina y Glicina que tienen unos microambientes muy característicos y únicos que los hacen más fácil de distinguir del resto (F3). Con dicha red fueron capaces de identificar aminoácidos propensos a mutar y mediante experimentos lograron observar que cada una de estas mutaciones tienen un aporte individual y positivo en la función de diferentes proteínas que analizaron, pero si combinas estas mutaciones, puedes generar un efecto aditivo en su función! Por ejemplo, usando todas las mutaciones identificadas para una proteína azul-fluorescente lograron mejorar su luminiscencia hasta 6 veces. En principio, un modelo como este permite operar con las mutaciones como si fueran sumas para mejorar sus funciones.
Refs:
