Post 9: LOGAN: el trabajo bioinformático más grande del año ✏️
Published:
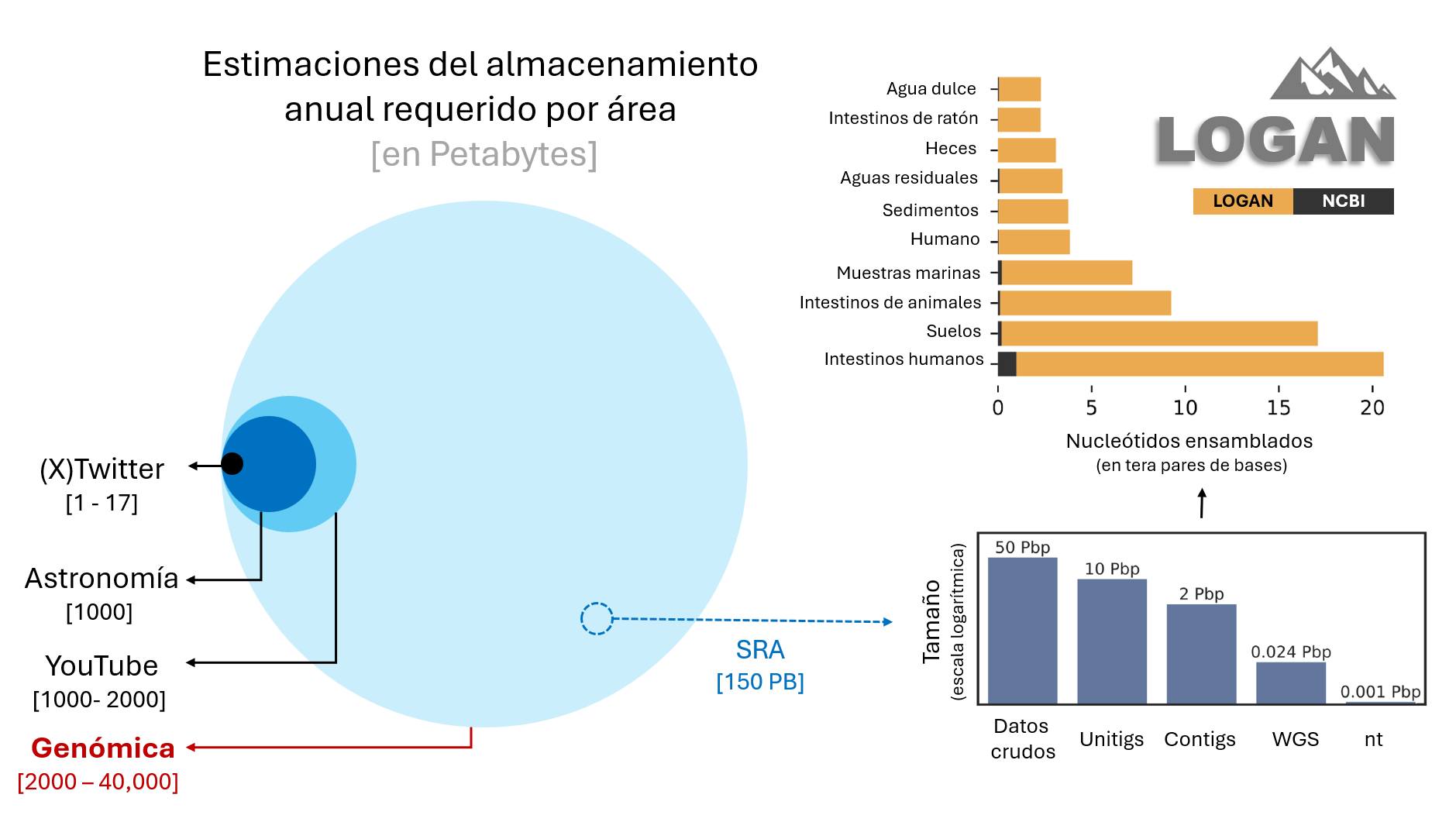
Usualmente cuando se trata de cosas grandes se habla de escalas astronómicas. Sin embargo, lo realmente grande en el mundo de los datos es la genómica, incluso más que Twitter o YouTube. Alrededor del mundo hay ~1000 laboratorios que pueden secuenciar ADN y cada uno produce mucha información anualmente.
El ADN secuenciado se guarda en muchas bases de datos. Una de ellas se llama SRA, y almacena datos crudos provenientes de humanos, animales o microbios. Al analizar la SRA solemos rastrear su origen para delimitar el conjunto que analizaremos, por ejemplo, todas las muestras de ratón. Aunque lo ideal sería poder procesar toda la información junta. Esto es lo que el proyecto LOGAN hizo.
En LOGAN procesaron el 96% de la SRA usando el computo en la nube de Amazon. El computo necesario para eso es tan masivo que equivale a ~2 millones de computadoras normales, y si una sola computadora hiciera todo tardaria unos ~3400 años! Para hacer eso, diseñaron una nube capaz de escalar con los datos, y dado que las secuencias de ADN eran tantas tuvieron que organizarlas (ensamblarlas) en piezas más grandes a las que llamaron Unitigs. Después ensamblaron los Unitigs en fragmentos aún más grandes de ADN llamados Contigs.
Se prefiere trabajar con información estructurada como los Contigs, a partir de los cuales podemos rastrear el origen taxonómico del ADN o mapear las proteínas que codifican. Al evaluar cuanta nueva información representa LOGAN respecto a la base de datos más famosa llamada NCBI (que se pude subdividir en otras bases como la nt o WGS) se encontró un aumento de hasta decenas de veces. Por ejemplo, los datos del microbioma humano aumentaron ~20 veces. Y toda esta información esta disponible para todos!
Seguramente con LOGAN encontraremos nueva biología, y posiblemente, se entrenaran futuras IAs tan grandes como la que usa ChatGPT (osea, GPT-4).
Refs:
