Post 3: Una base de datos taxogenómica 💻
Published:
La secuenciación masiva del DNA nos ha permitido conocer procariontes que son “imposibles” de cultivar. Todos esos genomas están disponibles en bases de datos como la NCBI o la GTDB, pero afrontamos un problema cada que encontramos un nuevo genoma: ¿Que chingados es este bicho?
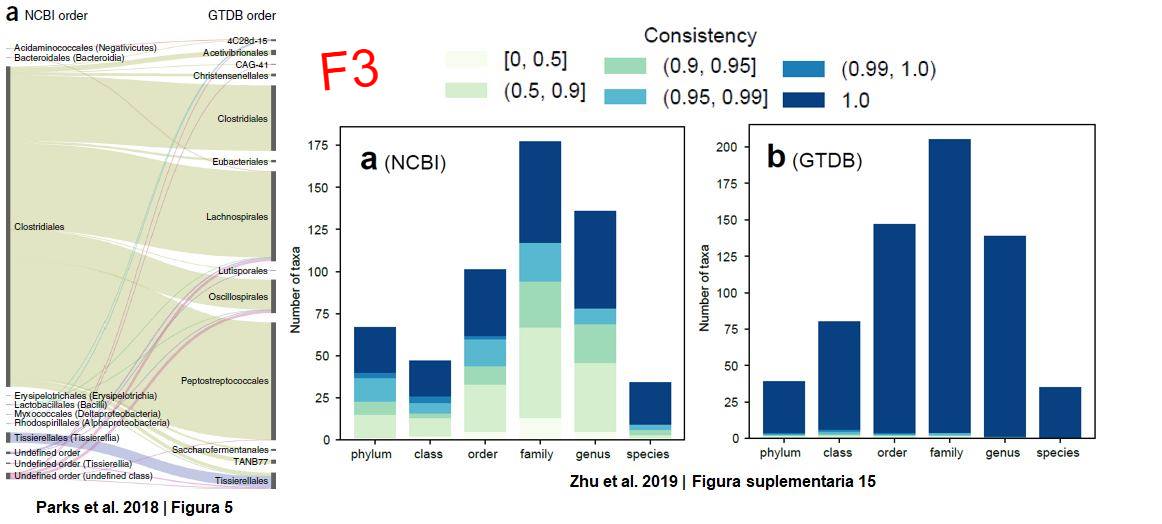
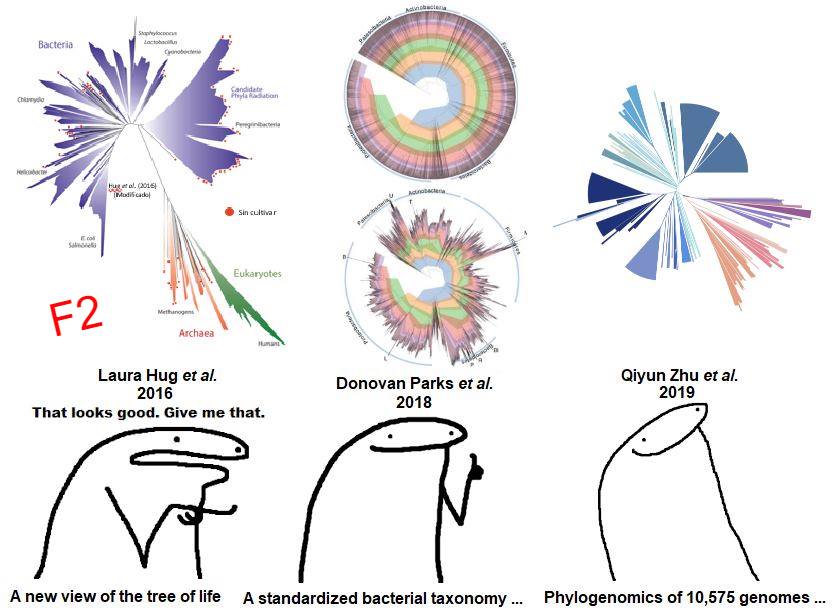
La taxonomía de la NCBI utiliza un enfoque polifásico que se basa únicamente pruebas experimentales. Sin embargo, ha demostrado tener muchos grupos de procariontes con relaciones taxonómicas no resultas (grupos polifiléticos) y no incluye toda la diversidad de microorganismos que se ha descubierto con la secuenciación masiva, lo cual podría llevar a conclusiones erróneas respecto a la clasificación de los organismos (F3). El problema aquí es: ¿qué recurso emplear para poder clasificar todas las formas de vida? (Figura 1 = F1 en las imágenes).

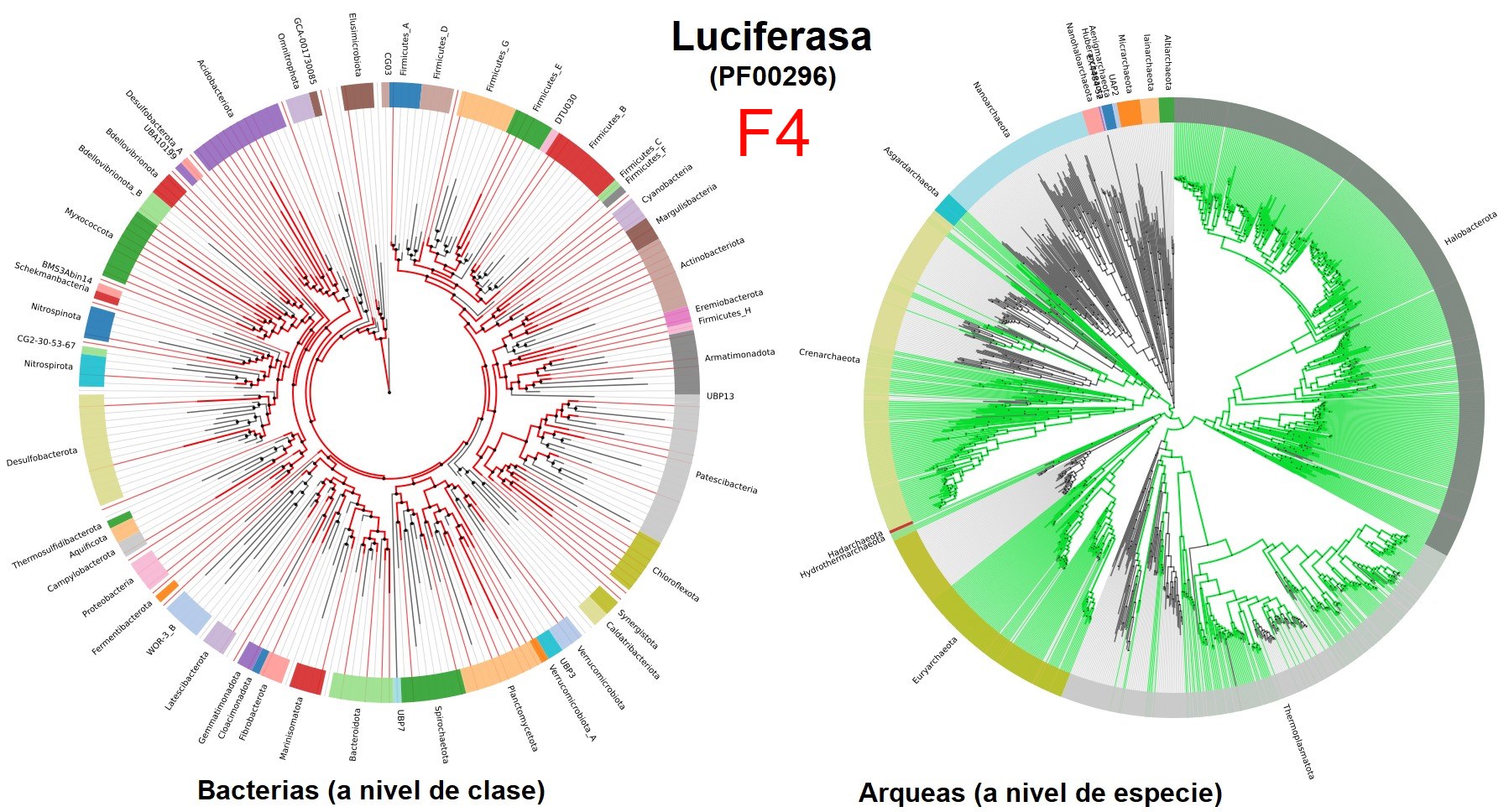
Laura Hug et al. demuestran que, con 16 genes de copia única, en su mayoría genes ribosomales, es posible clasificar los genomas de procariontes y eucariontes. Sin embargo, no abordaron el problema de relaciones polifiléticas de procariontes. En 2018 Parks et al. presentaron la GTDB, donde usando 120 y 122 genes de copia única para bacterias y arqueas, respectivamente, es posible clasificar a la gran mayoría de los procariontes de una forma mas precisa que las dos anteriores. Considero que la GTDB es una base de batos muy bien hecha y que además tiene varias herramientas muy útiles como la página de AnnoTree donde cualquiera con conocimientos básicos de biología y un poco de genómica puede buscar genes en todas las especies de procariontes conocidas hasta ahora, por ejemplo con el dominio de la enzima luciferasa famosa por ser responsable de la bioluminiscencia en varios bichos (F4). Sin embargo, una de sus mayores criticas es que se tuvieron que mover muchos de los grupos con relaciones polifileticas en la NCBI aunado a la idea de intentar estandarizar a la biodiversidad con parámetros biomatemáticos (F3).
En Diciembre de este año, Zhu et al. presentan uno de los trabajos mas robustos que he leído hasta ahora en donde usando 381 genes de copia única refuerzan la propuesta de que es necesario actualizar la NCBI o bien, usar clasificaciones como la GTDB o la WoL (Reference Phylogeny for Bacterial and Archaeal Genomes) desarrollada en su trabajo y que de hecho, resulta bastante congruente con las clasificaciones propuestas por la GTDB (F3, notece el indice de consistencia donde entre mas cercano a 1 significa una mayor congruencia en las clasificaciones). Sin embargo, la principal conclusión del trabajo es que, usando este conjunto de genes, las relaciones entre bacterias y arqueas cambia, pasando de ser organismos con una distancia filogenética considerable como se puede ver en la separación de las ramas del árbol de Laura Hug et al., a ser organismos que en realidad están mas cerca de lo que se pensaba, una idea de cambia lo que hemos creído durante ~12 años. Esto se debe a que aparentemente los genes ribosomales por si solos parecen estar sujetos a mayores fuerzas evolutivas que el resto de los genes entre arqueas y bacterias.
En resumen, aún no sabemos qué onda con como clasificar la vida, pero lo que si sabemos que el América puede pasar a chingar a su madre >:v
Refs:
