Post 7: Problemas serios en la taxonómia de procariontes 🔬
Published:
Actualmente, los microbiólogos tenemos problemas serios respecto a la taxonómia de procariontes.
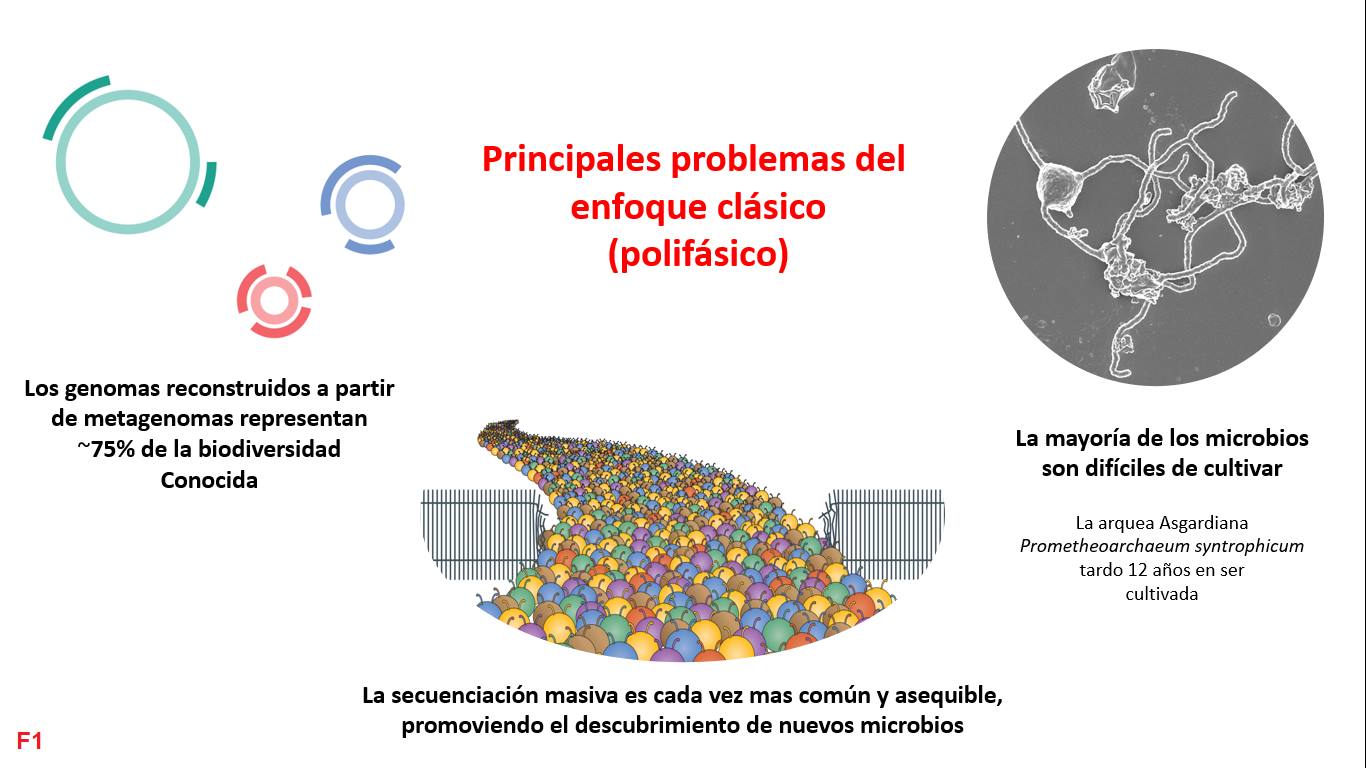
La forma clásica de reportar una nueva especie implica cultivarla en el laboratorio y realizar experimentos para conocer su metabolismo. Además, es necesario identificar su taxonomía, para ello, se suele usar el gen ribosomal 16S o genes marcadores. Finalmente, y siguiendo el código de nomenclatura, asignas un nombre y escribes un artículo con la propuesta, el cual se revisa, valida y acepta una vez que depositas tu cultivo en al menos dos colecciones internacionales. El problema con este proceso es que ya no es apto para la era genómica, pues la gran mayoría de las especies conocidas no se pueden cultivar (hasta ahora), algunas especies tardan años cultivarse y, además, cada vez es más fácil encontrar nuevas especies por secuenciación masiva (metagenómica) [F1].
En 2016, se hizo la propuesta formal de aceptar a los genomas como ‘referencia’ para nombrar a las especies, en lugar de usar el método usado hasta ahora que implica el cultivo (método polifásico). Esto dio paso a una gran discusión respecto a si era mejor unificar todos los microbios en un solo código, o bien, tener un código para cada tipo (cultivados y no cultivados).
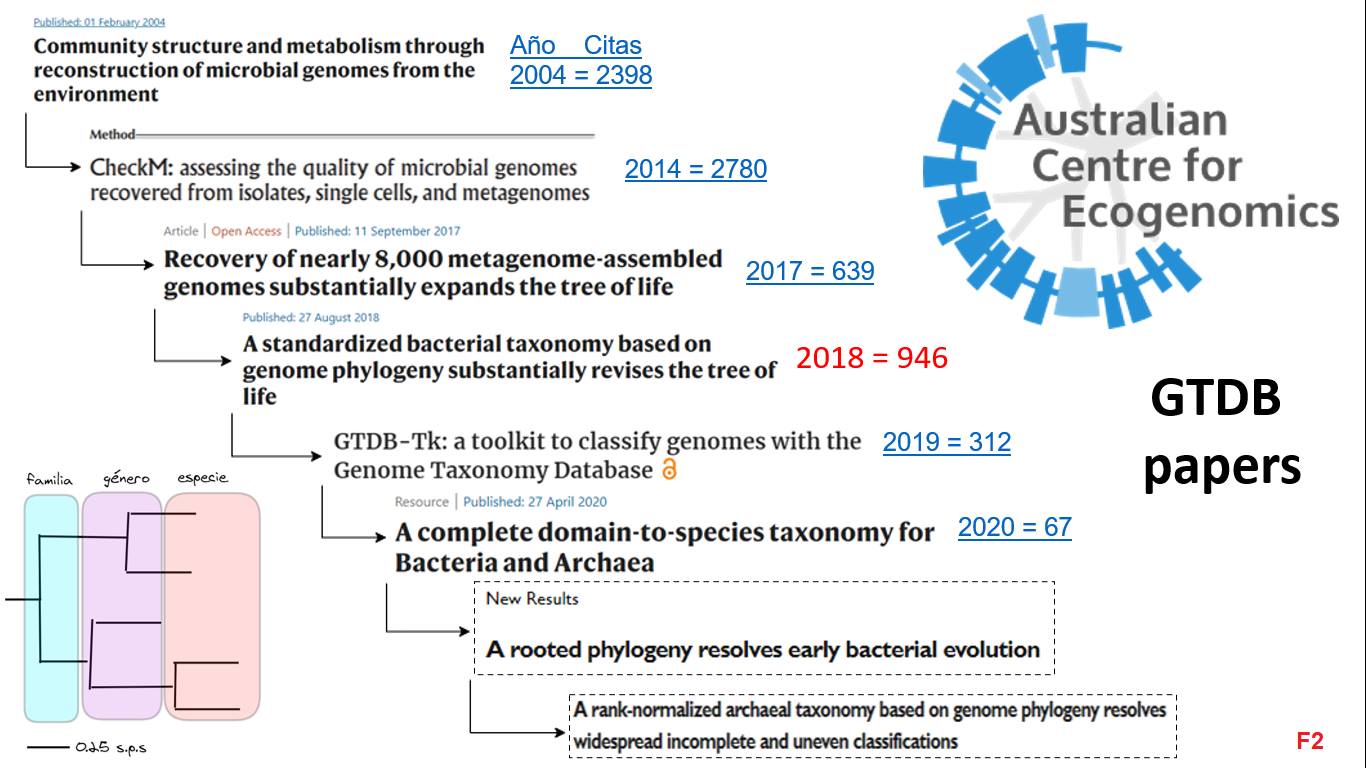
Desde los 60’s se propuso que las categorías taxonómicas (Familia, Genero, etc.) deberían ser asignadas considerando la historia evolutiva de los microbios (filogenia). Esta y otras ideas más fueron retomadas por científicos australianos en 2018 para proponer una taxonomía basada en genomas de procariontes conocida como GTDB. Para entenderla bien es necesario leer varios artículos [F2], pero en resumen: la conclusión es que muchos de los grupos de microbios están mal clasificados y además, considerando a los microbios sin cultivar, propusieron reconfigurarlos en ~32,000 especies de bacterias y arqueas que conocemos mediante su genoma.

En 2019 un grupo independiente de científicos llegaron a la misma conclusión: la taxonomía clásica tiene muchas inconsistencias y necesita actualizarse; además usando métodos distintos observaron consistencia en sus resultados respecto al grupo australiano. Aún más, los métodos desarrollados parecen ser útiles para analizar la historia evolutiva de los hongos, un grupo claramente diferente a los procariontes, respaldando la robustez del análisis bioinformático [F3]
Pese a todo, la propuesta de actualizar el código de nomenclatura fue rechazada apenas el año pasado. Siendo algunas de las razones el hecho de que los genomas de microbios sin cultivar son propensos a errores, que no sabemos para que sirven la mayoría de los genes y que la gran mayoría de microbios carecen de datos experimentales que respalden su fisiología. Lo cual ha llevado a discutir muchos de los demonios dentro de la comunidad de microbiólogos [F4].

¿Por qué importa esto? Porque tener claro con que grupo de microbios se está trabajando es calve para las conclusiones de las investigaciones y permite una comunicación clara. Personalmente, yo me inclino más por los métodos basados en genomas, pero que voy a saber si aún no tengo mi título de biólogo :’v
