Una guía para el diseño de proteínas basado en IA
Curso, Youtube, 2026
Hice este curso gratuito de 10 clases para introducirte al diseño de proteínas.
Contenido
- Descripción general
- Organización de clases
- Acceso a las diapositivas
- Cómo apoyar este proyecto
- Sobre mí
Note: there is a version of this page in English / Nota: aquí hay una versión en inglés de esta página
Descripción general
Quiero que más personas aprendan a diseñar proteínas usando Inteligencia Artificial (IA). Sin embargo, he encontrado tres problemas:
- hay mucha información y no es claro por dónde empezar a aprender ni cuáles son los temas necesarios que abarcar
- no hay cursos integrales en línea y en Español sobre el tema
- los cursos relacionados con el tema suelen ser costosos para la mayoría de los estudiantes de Latinoamérica
Para atender estos problemas, diseñé este curso gratuito de 37 horas, distribuido en 10 clases, para introducirte al diseño de proteínas basado en IA. El curso cuenta con dos recursos principales:
- Las 10 clases en YouTube
- Un repositorio en GitHub con los siguientes recursos:
- Herramientas: librerías organizadas en 25 categorías
- Recursos de aprendizaje: cursos, tutoriales y publicaciones útiles organizadas en nueve categorías
- Bases de datos: recursos para descargar datos genómicos y de proteínas organizados en 12 categorías
- Clases: enlaces a cada una de las clases y para descargar sus diapositivas
- Youtube: canales y videos recomendados para aprender sobre proteínas, matemáticas y ciencia de datos
Organización de clases
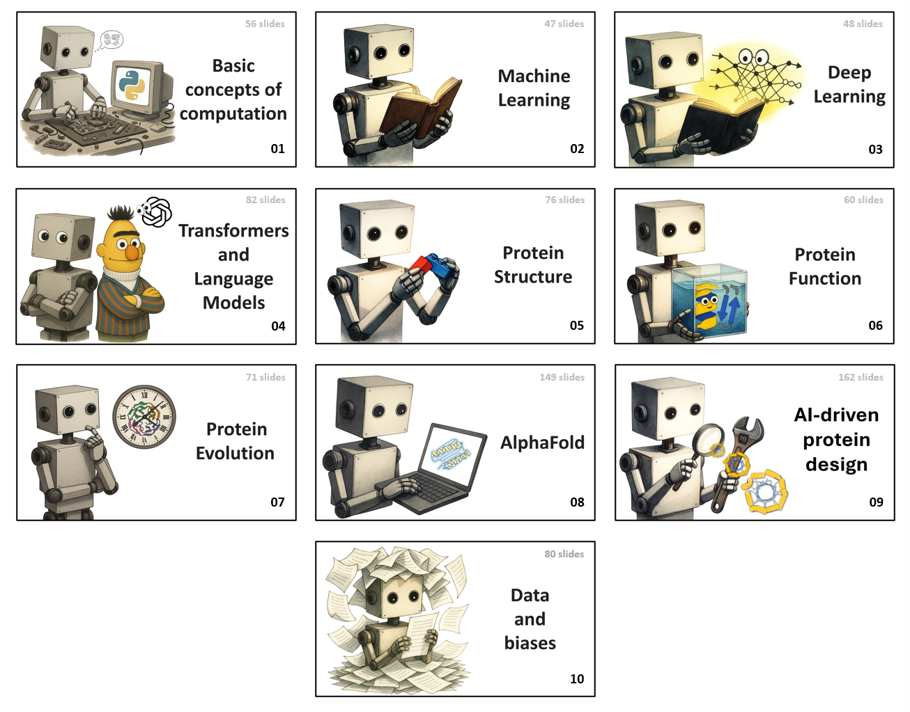
Las clases están organizadas de la 01 a la 10 para facilitar la comprensión de los temas. Por ejemplo, para revisar AlphaFold se requieren conceptos de biología estructural y deep learning que son explorados a detalle en sus respectivas clases. A continuación se describen brevemente las clases y sus temas:
- Conceptos básicos de computación: cómo funcionan las CPU y GPU, así como el software esencial para el análisis de datos.
- ¿Dónde comienza tu camino?
- Hardware
- CPU
- GPU
- Software
- Linux/Bash y GitHub
- Python
- Machine learning: qué es la IA y sus subcampos, las capacidades actuales de los algoritmos y cómo se entrena un modelo.
- Estado actual de la IA
- Cómo aprende la IA
- Patrones
- Operaciones de aprendizaje automático (MLOps)
- Paradigmas de aprendizaje
- Cómo entrenar un modelo
- Procesamiento de datos
- Cómo elegir un modelo
- Proceso de entrenamiento
- Deep learning: cómo funcionan las redes neuronales, los diferentes tipos de arquitecturas y el software utilizado para trabajar con ellas.
- Redes neuronales
- Neuronas
- Aprendizaje profundo
- Funciones de pérdida
- Retropropagación
- Optimizadores
- Arquitecturas
- Explicabilidad (por qué) e Interpretabilidad (cómo)
- Librerias de aprendizaje profundo
- Redes neuronales
- Transformers y modelos de lenguaje: funcionamiento de los Transformers y los modelos de lenguaje modernos.
- Modelos de lenguaje
- Transformers
- Arquitectura original
- Arquitecturas BERT y GPT
- Leyes de escala
- Pre-entrenamiento y post-entrenamiento
- Aprendizaje por refuerzo
- Desempeño y generalización
- Saturación de benchmarks
- Expectativas excesivas
- Cómo trabajar con LLMs
- Técnicas de optimización
- HuggingFace y Software 2.0
- Estructura de proteínas: principios de biología estructural y organización.
- Organización estructural
- Aminoácidos
- Estructura secundaria y terciaria
- Experimentos para la determinación de estructuras
- Visualizadores de estructuras
- Clasificaciones
- Plegamientos y dominios
- Primeros esquemas de clasificación
- Métricas de similitud
- Divergencia de secuencias y estructuras
- Esquemas de clasificación actuales
- La forma del universo proteico
- Distribución desigual
- Relaciones homólogas complejas
- Cambio de plegamiento
- Organización estructural
- Función de las proteínas: cómo adoptan su estructura y cómo se regula su función.
- Plegamiento de proteínas
- Entorno celular
- Termodinámica y entropía conformacional
- Función de las proteínas
- Difusión
- Dinámica molecular y funciones de energía
- Enzimas
- Anotación funcional
- Regulación funcional
- Alosterismo
- Regulación transcripcional
- Modificaciones post-traduccionales
- Proteoestasis y fisiología del huésped
- Plegamiento de proteínas
- Evolución de proteínas: origen y diversificación a partir de péptidos simples.
- Niveles de organización biológica
- Evolución a través de escalas espacio-temporales
- Evolución química
- Evolución biológica
- Hipótesis del mundo de ARN y evolución del ribosoma
- Proteínas ancestrales
- Diversificación
- El espacio de secuencia
- Mutaciones
- Robustez, evolubilidad y promiscuidad
- Evolución de la función proteica
- Epistasis: Cómo las interacciones moldean la evolución
- Interacciones residuo-residuo y proteína-proteína
- Aleatoriedad de las mutaciones
- Niveles de organización biológica
- AlphaFold: visión general de las arquitecturas AF2 y AF3 y su impacto.
- El impacto de AlphaFold
- AlphaFoldmania
- Predicción de estructuras antes de AlphaFold
- AlphaFold
- AlphaFold2
- Modelos de lenguaje de proteínas
- Arquitectura
- La era post-AlphaFold2
- AlphaFold3
- Modelos de difusión para modelado macromolecular
- Arquitectura
- La era post-AlphaFold3
- El impacto de AlphaFold
- Diseño de proteínas basado en IA: paradigmas del diseño de proteínas e implementaciones modernas basadas en IA.
- Diseño de proteínas
- La IA en el mercado biotecnológico
- Avances desde métodos clásicos a métodos basados en IA
- Consideraciones básicas para aumentar el éxito de un diseño
- Diseño racional
- Enfoques experimentales y bioinformáticos clásicos
- Modelado macromolecular y recombinación
- Diseño evolutivo
- Evolución dirigida, reconstrucción de secuencias ancestrales y diseño por consenso
- Aprendizaje de representaciones
- Representaciones (macro)moleculares y Foldseek
- Modelos de lenguaje de proteínas y ESMFold
- Explicabilidad e interpretabilidad de modelos de lenguaje de proteínas
- Leyes de escala y multimodalidad en modelos de lenguaje de proteínas
- IA Generativa
- Integración de datos multimodales
- Generación de secuencias
- Generalización y predicción de aptitud con modelos de lenguaje
- Plegamiento inverso y ProteinMPNN
- Generación de estructuras con modelos de difusión
- Selección de modelos y puntuación computacional de candidatos
- Generalización de modelos y datos sintéticos
- Resumen
- Diseño de proteínas
- Datos y sesgos: Bases de datos, técnicas de procesamiento, sesgos inherentes a los datos y generalización aplicada a la biología
- Los datos masivos son ómicos.
- Propiedades de un buen conjunto de datos
- Principales bases de datos
- PDB
- UniProt
- Conjuntos de datos del NCBI
- Otras bases de datos de interés
- Procesamiento de datos
- Limpieza de datos en biología
- Herramientas básicas para manipulación de datos biológicos
- División de datos
- Generalización en biología (de proteínas)
- Fuga de datos y otros problemas inherentes
- Sesgos en los datos
- Una guía para el diseño de proteínas basado en IA
- Los datos masivos son ómicos.
Acceso a las diapositivas
Este curso se compone de +800 diapositivas con las fuentes de las imágenes y citas, así como recursos recomendados para profundizar en cada tema, en la sección de notas. Recomiendo revisar las diapositivas con PowerPoint. Puedes descargar las diapositivas en Zenodo (links directos a descarga) y Google Drive:
| Tema | Diapositivas | YouTube |
|---|---|---|
| Conceptos básicos de computación | Drive, Zenodo | Video |
| Machine learning | Drive, Zenodo | Video |
| Deep learning | Drive, Zenodo | Video |
| Transformers y modelos de lenguaje | Drive, Zenodo | Video |
| Estructura de proteínas | Drive, Zenodo | Video |
| Función de proteínas | Drive, Zenodo | Video |
| Evolución de proteínas | Drive, Zenodo | Video |
| AlphaFold | Drive, Zenodo | Video |
| Diseño de proteínas basado en IA | Drive, Zenodo | Video |
| Datos y sesgos | Drive, Zenodo | Video |
Al liberar las diapositivas pretendo que tengas acceso a información para profundizar. Si eres docente y has adoptado este material para tus clases, por favor, házmelo saber. Me encantaría conocer cómo mejoraste el curso y saber que más personas han aprendido sobre ciencia de proteínas.
Sin embargo, si identificas que alguien ha plagiado total o parcialmente este curso Y ADEMÁS cobra dinero por acceder a él, te agradecería que me lo notificaras, pues su elaboración me llevó mucho tiempo y trabajo, y el plagio es una seria falta de profesionalismo y ética.
Cómo apoyar este proyecto
Si este curso te fue útil y deseas retribuirlo económicamente, puedes hacer una donación vía PayPal. Las donaciones pueden ser de cualquier monto o de 12, 30 o 45 dólares estadounidenses (sugerencias basadas en la economía de los estudiantes de Latinoamérica). Haz clic en la siguiente imagen si quieres donar.
Si no cuentas con gran libertad económica, pero quieres expresar tu gratitud, puedes enviarme tus comentarios a mi correo: gamamiguelangel@gmail.com
Finalmente, te agradecería que compartieras este curso con colegas interesados, o bien, que difundieras (dar repost) el anuncio oficial de este curso en distintas redes sociales:
Sobre mí
Soy Miguel Angel González Arias. Soy un biólogo mexicano y me gustan las proteínas, los microbios y la computación. Para más detalles sobre mi, redes sociales y otros medios de contacto, consulta la siguiente pagina:

