Post 11: Filogenias masivas de microbios 🧬
Published:
Hoy es el día del microbioma! Así que resumiré mi artículo favorito sobre evolución de microbios.
El equipo de Qiyun se dio a la tarea de analizar el árbol de la vida de los microbios. Esto no es fácil, pues aunque tenemos muchos genomas, muchos están incompletos y tampoco es claro como identificar a los mejores.
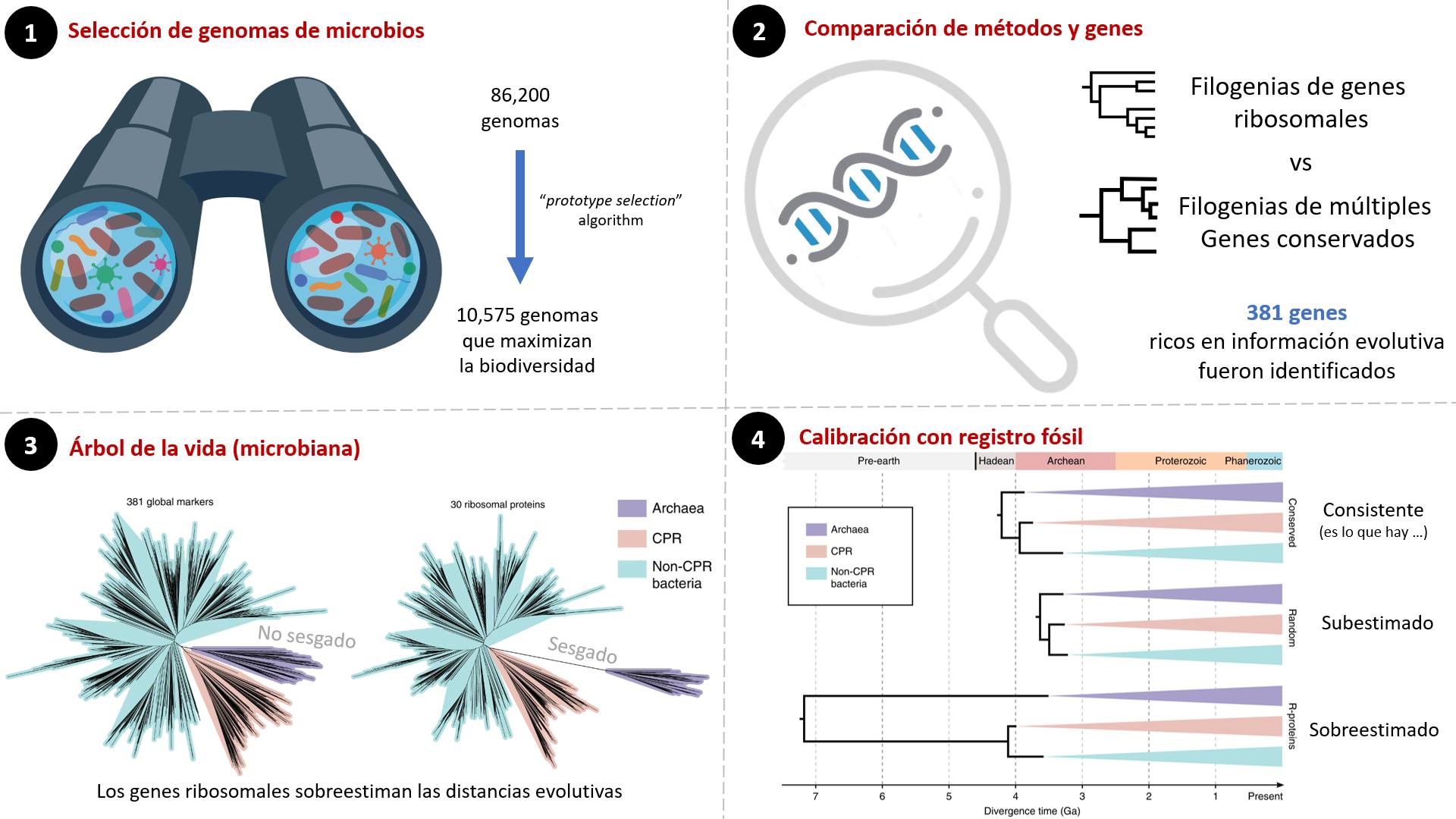
1) Para identificar genomas representativos, escribieron un algoritmo que busca maximizar la biodiversidad a partir de las distancias genéticas entre microbios y encontraron 10,575 genomas representativos.
2) El siguiente paso es identificar genes ricos en información evolutiva a partir de estos genomas. Después de varios análisis, identificaron 381 genes con esta condición. Y con ellos, compararon muchos métodos de inferencia filogenética.
3) Con estos genes ricos en información pudieron reconstruir el árbol de la vida de los microbios. Observaron que, si usamos solo proteínas del ribosoma, las relaciones evolutivas entre bacterias y arqueas se sobrestiman.
4) Finalmente, a este árbol de la vida le añadieron información del registro fósil para estimar los tiempos de divergencia de muchos grupos de microbios. Observaron que si usan proteínas ribosomales, estos tiempos no son consistentes con las estimaciones del origen de la vida. Si toman genes al azar, los tiempos de divergencia son subestimados. Pero si toman los 381 genes se obtiene una muy buena consistencia con lo que sabemos sobre evolución biológica y geológica.
No es un articulo facil de leer, pero si valioso. Y hasta viene con pagina web https://biocore.github.io/wol/
Refs:
